生命科学学院庞尔丽教授课题组在Horticulture Research发表预测植物基因组潜在顺式调控元件的新方法
2023年2月28日,庞尔丽教授课题组在Horticulture Research(IF2022=7.3)发表题为“Identification of Clade-wide Putative Cis-regulatory Elements from Conserved Non-coding Sequences in Cucurbitaceae Genomes”的研究论文,以葫芦科物种为例阐述了一种新的预测植物基因组中保守非编码顺式调控元件的方法。
植物经过长期环境驯化,演化出了复杂而精细的基因时空表达调控机制来应对不同的生境挑战。作为基因表达调控的重要角色之一,顺式调控元件(cis-regulatory element,CRE)是一类主要调节邻近基因表达的非编码DNA区域。CRE一般只有几个到几十个碱基大小,常常分布在紧邻基因的上下游或是基因的内含子区,有时也会远离基因区甚至跨越不同染色体。研究表明一些保守的非编码序列(conserved noncoding sequences, CNS)属于顺式调控元件,在脊椎动物等多个不同支系中发现它们常常参与胚胎发育等关键生物学过程的调控。鉴于植物基因组的复杂性,比如全基因组加倍事件、频繁的基因组重组和高比例的重复序列等,使得从一组进化关系紧密相关的多个基因组中预测识别该支系范围内存在的具有顺式调控功能的保守非编码元件面临挑战。
本研究以葫芦科中染色体水平基因组组装的12个物种为例,提出了一种基于全基因组比对锚点(alignment anchor)作为基因组标记的共线性识别策略,利用该策略首先识别出共线性区域,接着采用phastCons模型从12-way的全基因组比对结果中预测得到高度保守的非编码序列,然后基于保守的非编码序列及其周围编码蛋白基因的共线性关系预测了潜在的顺式调控保守非编码元件(cis-regulatory conserved noncoding element, cisRCNE)(图1)。在12个葫芦科物种中,一共预测到了632,112个保守的非编码序列,其中在黄瓜基因组中预测了3,271个潜在的顺式调控保守非编码元件和基因调控对。通过整合分析黄瓜果实发育三个阶段的RNA-seq和ChIP-seq测序数据,使得其中的98个顺式调控非编码元件和基因调控对得到了调控关系的验证,同时发现部分cisRCNEs有可能参与了黄瓜果实发育过程由H3K27me3甲基化修饰介导的基因表达调控过程(图2)。
这是首次在葫芦科中报道的CNSs和cisRCNEs相关研究结果。本研究预测的葫芦科CNSs、cisRCNEs及其潜在的调控靶基因都通过数据库形式公开发布在http://cmb.bnu.edu.cn/cisRCNEs_cucurbit/上,这些材料是未来开展葫芦科物种中基因组功能研究工作的重要资源。
本研究同时提出了一种识别植物某一类进化支系范围内cisRCNE的计算框架(图3)。为了验证整个预测方法的稳健性,本研究还在6个染色体水平组装的茄科植物中开展了类似工作,均得到了稳定且相似的结果(https://cmb.bnu.edu.cn/cisRCNEs_solanaceae/)。这是首次基于序列保守性和调控关系共线性两方面信息的指导,在全基因组范围内从CNSs中预测识别cisRCNEs的一种计算策略,为植物比较基因组研究提供了新思路。
北京师范大学生命科学学院生命科学与技术国家级实验教学示范中心的宋宏涛博士为论文第一作者,生物多样性与生态工程教育部重点实验室的庞尔丽教授为该论文的通讯作者,在读研究生王琪参与了研究,林魁教授和青岛农业大学园艺学院的张忠华教授参与了指导。该研究得到了国家自然科学基金的资助。
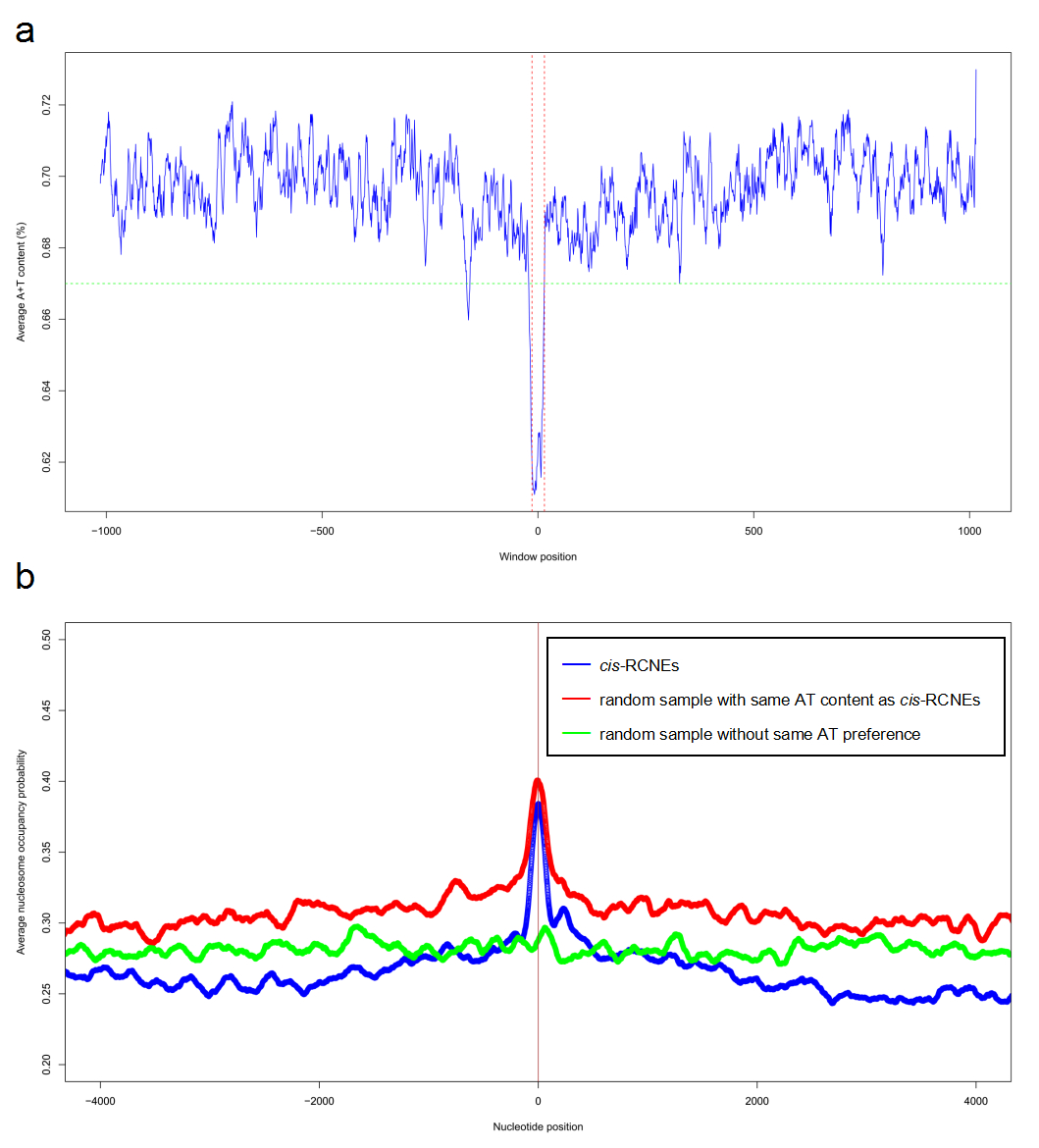
图1 cisRCNEs的AT-drop序列特征及核小体高占位趋势
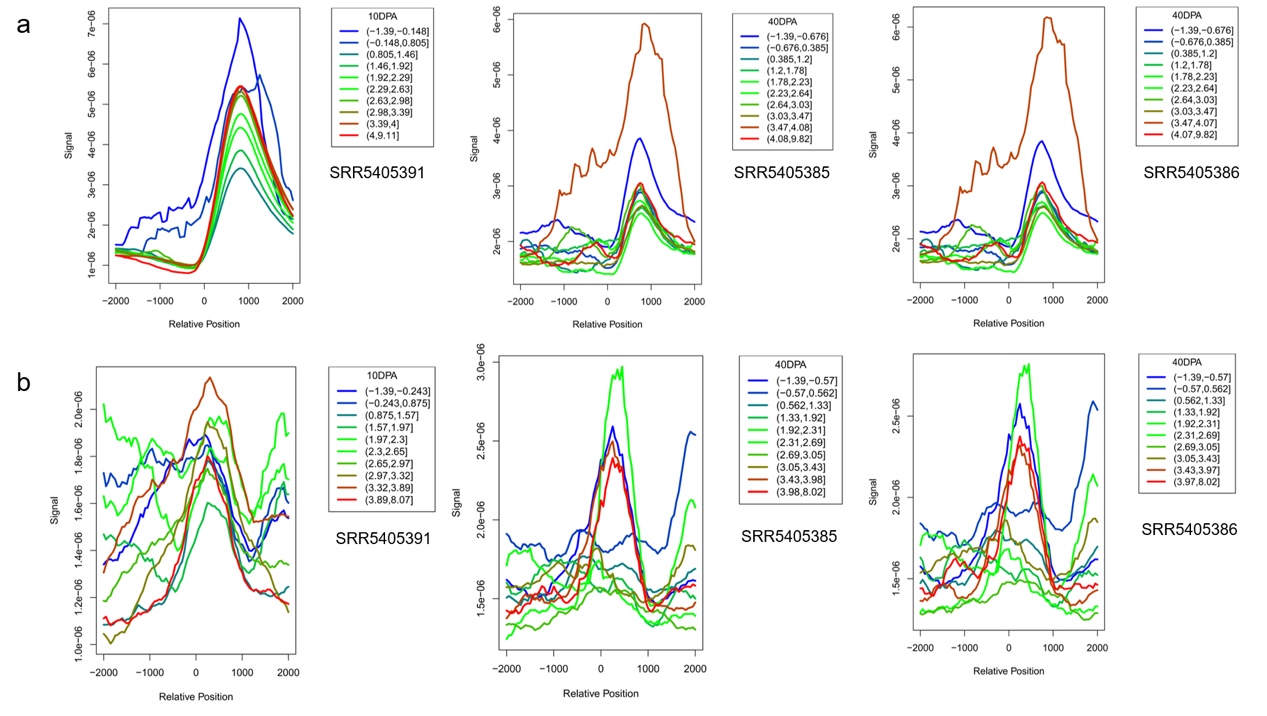
图2 黄瓜两个果实发育阶段中出现启动子(上)和cisRCNEs(下)区的H3K27me3修饰强信号
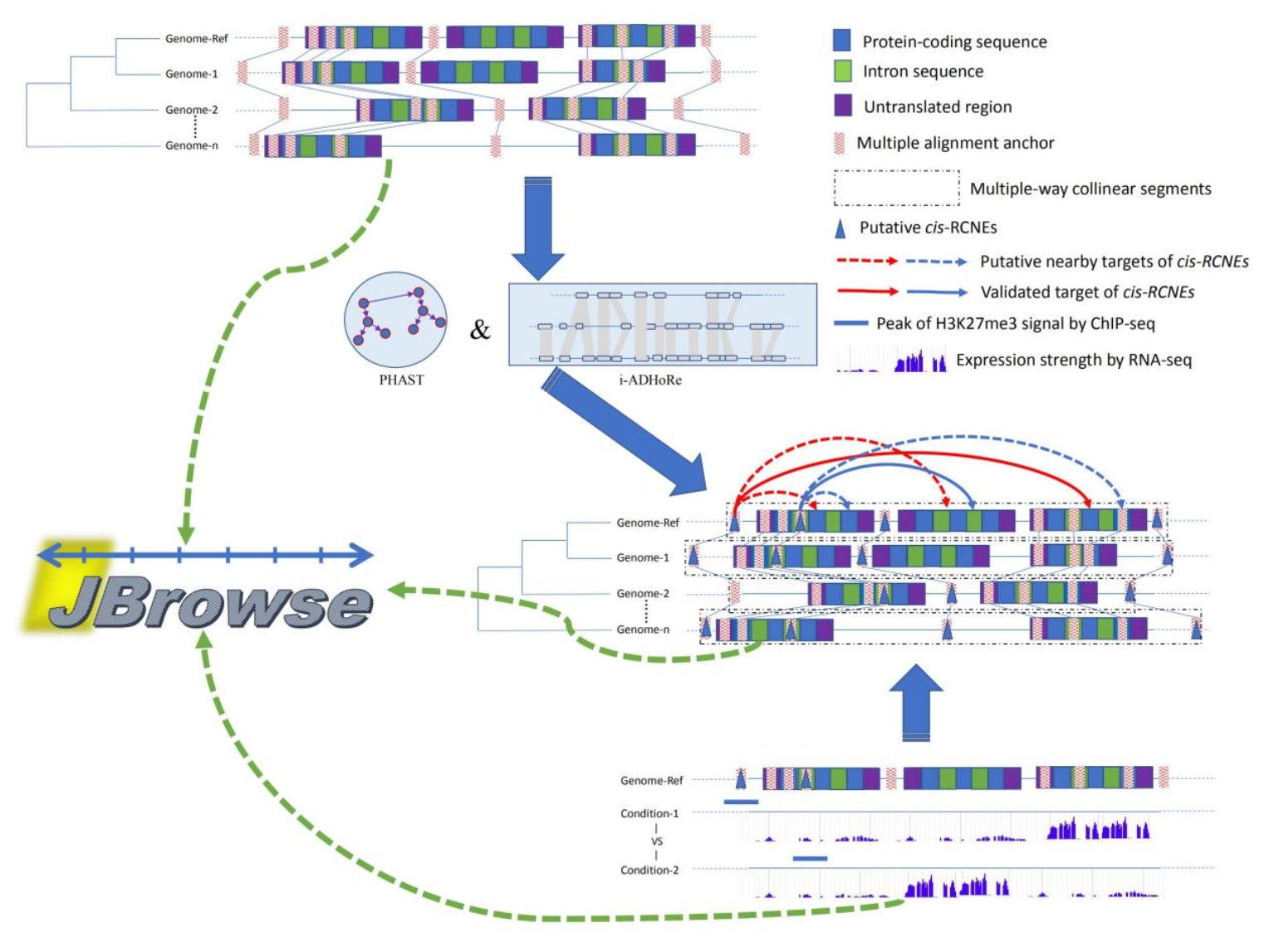
图3 计算框架图
原文链接:https://academic.oup.com/hr/advance-article/doi/10.1093/hr/uhad038/7060410
